A informação baseada em electroforese foi disposta em 3 tabelas: A tabela ELECTSTUDIES (Estudos de electroforese), que nos dá uma imagem dos estudos conduzidos em diferentes populações de certas espécies; a tabela ELECTDAT (dados de electroforese) que nos dá os loci que foram sondados em certos estudos; e a tabela ELECTSUB que contém os alelos que foram detectados em certos loci.
No seu conjunto, as tabelas informam sobre a estrutura e variabilidade genética de populações naturais e cultivadas. Isto é importante para a selecção de espécies/raças para a aquacultura e ajuda em programas de gestão e conservação de stocks naturais.
Conforme mais dados vão entrando nesta tabela, será possível identificar falhas na investigação (i.e., poucos estudos em espécies importantes) e os métodos mais apropriados para a caracterização genética de várias espécies.
As tabelas contêm frequências alélicas de estudos de electroforese de populações de peixes, tanto selvagens como de cultivo. Também contêm informação sobre enzimas, número total de loci estudados, tecidos e sistema tampão utilizados, valores de heterozigotia e proporções de loci polimórficos. Os campos das tabelas são:
Localidade e País: referem o local onde os espécimes foram recolhidos.
Nº total de loci: Este campo refere o nº de loci examinados.
Heterozigotia observada: é a proporção de indivíduos, numa população, que são heterozigóticos num determinado número de loci. Um indivíduo com 2 alelos diferentes num locus particular é chamado heterozigótico. Um indivíduo é chamado homozigótico, quando dois alelos num locus particular são iguais.
Heterozigotia esperada: é por outro lado, a proporção de indivíduos que são heterozigóticos esperados, baseados nas frequências alélicas e assumindo o equilíbrio de Hardy-Weinberg. Isto é computorizado para cada locus, população e espécie, e descreve por exemplo, o potencial para reprodução selectiva (Fig. 53).
Loci polimórficos: refere-se ao número de loci que numa amostra são polimórficos a dividir pelo nº total de loci examinados (Fig. 54). Para homogeneizar os dados, o critério 95% é aqui utilizado, onde um locus é considerado polimórfico se a frequência do alelo mais comum não ultrapassa 0,95. Se os dados se referem ao critério 99%, isso será indicado no campo comentários.
Enzima: Este campo de escolha inclui nomes, abreviaturas e número recomendado de enzimas e outras proteínas, normalmente analisada em trabalhos de genética de peixes. Os nomes e número utilizados baseiam-se na nomenclatura recomendada pelo "International Union of Biochemistry's Nomenclature Committee" (Shaklee et al. 1990)
Locus: refere-se à localização ou posição específica de um gene no cromossoma. Um gene é um comprimento específico do ADN (ácido desoxirribonucleico) ocupado por um locus. Um locus é chamado monomórfico se apenas um alelo é conhecido e polimórfico quando existem dois ou mais alelos no locus. Quando dois ou mais loci estão envolvidos na produção de diferentes formas da mesma proteína (isoenzimas), o locus mais anodal é designado por 1, a seguir 2 e assim sucessivamente. Por vezes o locus é designado por letras, o mais anodal é designado por A, o seguinte B e assim sucessivamente.
Tecido: Refere-se ao tipo de amostra de tecido utilizado na electroforese. As escolhas disponíveis são: músculo esquelético; músculo visceral; coração; rim; fígado; sangue; muco; lente ocular; corpo inteiro; outras. A última escolha refere-se a tecidos que se encontram no campo comentários.
Método: é um campo de escolha que refere o tipo de electroforese utilizado. A electroforese em gel é um dos métodos mais frequentes para estudar a variação genética individual ao nível de raças e de espécies. Quatro escolhas são dadas: gel de amido; gel de poliacrilamido; sulfato dodecil de sódio; outros métodos.
tema tampãote campo refere-se ao sistema tampão electroforético utilizado para uma clara resolução de proteínas e enzimas específicas. Os 15 sistemas tampão mais utilizados estão descritos em Boyer et al. (1963); Ridgway et al. (1970), Shaw & Prasad (1970), Selander et al. (1971), e Clayton & Tretiak (1972).
pH: Refere-se à acidez ou basicidade do sistema tampão utilizado.
Amostra: este campo refere-se ao número de amostras estudadas por local ou por população.
Alelo: é uma das várias formas alternativas de um gene específico. Os alelos distinguem-se pelos produtos das suas proteínas (enzimas) durante a electroforese. A mobilidade electroforética relativa das enzimas num zimograma é expressa em termos numéricos. As mobilidades relativas são calculadas baseadas no alelo mais comum, que é considerado 100 (ou -100 para locus catódicos). O sinal negativo assinala um alelo com mobilidade catódica.
A Frequência alélica: de um determinado locus é calculada usando a seguinte fórmula: Frequência do alelo A = 2 . (frequência do genótipo AA) + (frequência do genótipo Aa)/2n, onde n= número de indivíduos estudados.
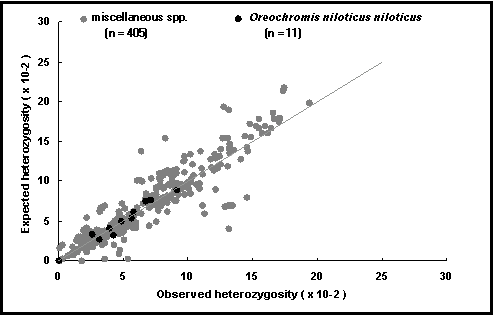
Fig. 53. Heterozigotia esperada vs observada para Oreochromis niloticus niloticus (pontos escuros) e para outras espécies. A linha representa proporções de 1 : 1 e identifica os pontos onde a heterozigotia esperada é igual à observada. Os valores acima desta linha podem resultar de consanguinidade os que estão bastante em baixo do cruzamento de raças.
Esta tabela contém mais de 11,000 registos (um registo representa alelos num simples locus,/I>) de frequências alélicas para mais de 900 estudos e para mais de 800 populações/raças de peixe. A actualização desta base de dados em colaboração e utilizando referências identificadas por Skibinski et al. (1991) fará desta tabela o mais longo repositório de dados sobre variabilidade genética de peixes.
Podem ser criados vários gráficos a partir desta tabela que mostram:
A linha de correspondência entre heterozigotia esperada e observada (Fig. 53): examina se a variabilidade genética (H e P) foi reduzida na populações em cativeiro em relação às populações em ambiente natural;
A relação entre conteúdo em ADN e ordem filogenética, de acordo com Fishes of the world (Nelson, 1994) (Fig. 51);
A relação entre o número de cromossomas e teor de ADN; e
A relação entre o teor de ADN e a razão aspecto da barbatana caudal (Fig. 52 e Caixa 28).
Todos estes gráficos podem ser acedidos através da tabela GENÉTICA, activando a espécie. Em alternativa, pode seleccioná-los no menú Gráficos em Relatórios.
As referências mais importantes utilizadas são Winans (1980), McAndrew & Majumder (1983), Macaranas et al. (1986, 1995), van der Bank et al. (1989) e Carvalho et al. (1991) Pouyaud & Agnèse (1995).
Atingir a cobertura completa das frequências alélicas e relatar a informação sobre peixes já publicada, é ainda um grande desafio, e envolve a resolução de problemas devido à falta de homogeneização entre publicações, o que impede ainda a associação de dados (Agustin et al. 1993, 1994).
Chega-se à tabela ELECSTUDIES clicando no botão Biologia na tabela ESPÉCIES, no botão Genética na tabela BIOLOGIA, depois no botão Frequência Alélica na janela GENÉTICA. Se clicar duas vezes sobre uma das linhas na LISTA DE ESTUDOS DE ELECTROFORESE DISPONÍVEIS pode obter informações relativas ao estudo designado.
Deste ponto, chega-se à tabela ELECDAT clicando no botão Dados De Electroforese. Uma lista de enzimas utilizadas aparece, e clicando numa enzima em particular, os detalhes sobre a mesma são fornecidos.
Chega-se à tabela ELECSUB clicando no botão Frequência Alélica na tabela INFORMAÇÃO DE LOCUS.
Nuestro agradecimiento para R.E. Brummett, A.E. Eknath, G.C. Mair, J.G. McGlade, D. Pauly, R.S.V. Pullin y D.O. Skibinski, por sus consejos acerca de la estructura y el contenido de esta tabla.
Agustin, L.Q., R. Froese, A.E. Eknath and R.S.V. Pullin. 1993. Documentation of genetic resources for aquaculture - the role of FishBase, p. 63-68. In D. Penman, N. Roongratri and B. McAndrew (eds.) International Workshop on Genetics in Aquaculture and Fisheries Management. ASEAN-EEC Aquaculture Development and Coordination Programme, Bangkok, Thailand.
Agustin, L.Q., M.L.D. Palomares and G.C. Mair. 1994. FishBase: a repository of genetic information on fish. Poster presented at the Fifth International Symposium on Genetics in Aquaculture, 19-25 June 1994, Dalhousie University, Halifax, Nova Scotia, Canada.
Boyer, S.H., D.C. Fainer and E.J. Watson-Williams. 1963. Lactate dehydrogenase variant from human blood: evidence for molecular subunits. Science 141:642-643.
Carvalho, G.R., P.W. Shaw, A.E. Magurran and B.H. Seghers. 1991. Marked genetic divergence revealed by allozymes among populations of the guppy Poecilia reticulata (Poeciliidae), in Trinidad. Biol. J. Linn. Soc. 42:389-405.
Clayton, J.W. and D.N. Tretiak. 1972. Amine-citrate buffers for pH control in starch gel electrophoresis. J. Fish. Res. Board Can. 29:1169-1172.
Macaranas, J.M., N. Taniguchi, M.J.R. Pante, J.B. Capili and R.S.V. Pullin. 1986. Electrophoretic evidence for extensive hybrid gene introgression into commercial Oreochromis niloticus (L.) stocks in the Philippines. Aquacult. Fish. Manage. 17:249-258.
Macaranas, J.M., L.Q. Agustin, M.C.A. Ablan, M.J.R. Pante, A.E. Eknath and R.S.V. Pullin. 1995. Genetic improvement of farmed tilapias: biochemical characterization of strain differences in Oreochromis niloticus. Aquaculture International 3:43-54.
McAndrew, B.J. and K.C. Majumdar. 1983. Tilapia stock identification using electrophoretic markers. Aquaculture 30:249-261.
Nelson, J.S. 1994. Fishes of the world. 3rd edition. John Wiley and Sons, New York. 600 p.
Pouyaud, L. and J.-F. Agnèse. 1995. Phylogenetic relationships between 21 species of three tilapiine genera Tilapia, Sarotherodon and Oreochromis using allozyme data. J. Fish Biol. 47(1):26-38.
Ridgway, G.J., S.W. Sherburne and R.D. Lewis. 1970. Polymorphism in the esterases of Atlantic herring. Trans. Am. Fish. Soc. 99:147-151.
Selander, R.K., M.H. Smith, S.Y. Yang, W.E. Johnson and J.B. Gentry. 1971. Biochemical polymorphism and systematics in the genus Peromyscus. I. Variation in the old field mouse (Peromyscus polionotus). Studies in Genetics VI. Univ. Texas Publ. 7103:49-90.
Shaklee, J.B., F.W. Allendorf, D.C. Morizot and G.S. Whitt. 1990. Gene nomenclature for protein-coding loci in fish. Trans. Am. Fish. Soc. 119:2-15.
Shaw, C.R. and R. Prasad. 1970. Starch gel electrophoresis of enzymes - a compilation of recipes. Biochem. Genet. 4:297-320.
Skibinski, D.O.F., M. Woodwark and R.D. Ward. 1991. The protein diversity database. University College of Swansea, Singleton Park, Swansea, Wales and CSIRO Division of Fisheries, Tasmania, Australia. 16 p.
van der Bank, F.H., W.S. Grant and J.T. Ferreira. 1989. Electrophoretically detectable genetic data for fifteen southern African cichlids. J. Fish Biol. 34:465-483.
Winans, G.A. 1980. Geographic variation in the milkfish Chanos chanos. I. Biochemical evidence. Evolution 34(3):558-574.
Christine Casal e Liza Agustin